Herramientas para visualizar modelos de estructuras tridimensionales

La estructura de una proteína o, más concretamente, la disposición tridimensional de los átomos en una molécula proteica, determina cómo interactúa la proteína con otras moléculas de la célula y cómo lleva a cabo sus funciones biológicas.
Herramientas en línea para visualizar estructuras tridimensionales de proteínas
El Banco de Datos de Proteínas (PDB, por sus siglas en inglés), es una base de datos que proporciona acceso gratuito a datos estructurales de grandes moléculas biológicas, como proteínas y ácidos nucleicos. Se considera un valioso recurso para los investigadores en el campo de la biología estructural.
PDB permite avances en la ciencia y la educación al proporcionar acceso y herramientas para la exploración, visualización y análisis de estos datos en el contexto de anotaciones externas que proporcionan una visión estructural de la biología.
Un Modelo de Estructura Computarizada (CSM, por sus siglas en inglés) es un modelo de estructura proteica generado mediante métodos computacionales como el modelado homológico o el modelado ab initio. Estos modelos se crean a partir de la secuencia aminoacídica de la proteína y otra información estructural disponible.
CSM es de gran utilidad cuando no se dispone de las estructuras experimentales o cuando éstas son de baja resolución. El sitio web RCSB PDB integra PDB con datos CSM procedentes de AlphaFold, lo que lo convierte en el sitio más extenso para renderizar en línea estructuras de proteínas en 3D (Imagen 1).
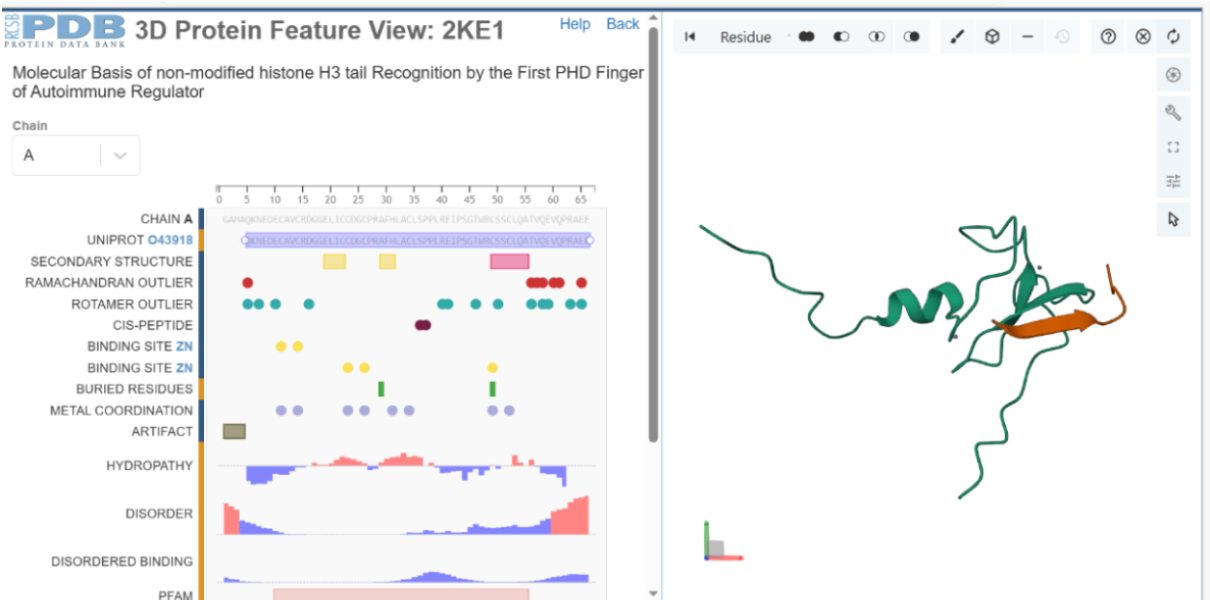
Image 1. Visualización de la estructura de una proteína en RCSB Protein Data Bank.
AlphaFold es un algoritmo de predicción de estructuras de proteínas basado en el deep learning y desarrollado por el equipo de DeepMind. Todos sus datos están disponibles gratuitamente tanto para uso académico como comercial (bajo una licencia CC-BY 4.0). El software es muy preciso en la predicción de estructuras de proteínas. Utiliza una red neuronal para generar la estructura 3D de una proteína a partir de su secuencia de aminoácidos.
Proporciona más de 200 millones de predicciones de estructuras de proteínas. Solo en 2021, una colaboración entre el Laboratorio Europeo de Biología Molecular y DeepMind ha predecido las estructuras de más de 350 000 proteínas de 21 organismos modelo.
Otro servicio en línea es UniProt, el cual proporciona un acceso completo y de alta calidad a secuencias de proteínas e información funcional. Se trata de una colaboración entre el Instituto Europeo de Bioinformática (EMBL-EBI), el Instituto Suizo de Bioinformática (SIB) y el Protein Information Resource (PIR).
Contiene una amplia y detallada documentación sobre la función biológica de las proteínas procedente de publicaciones científicas. Incluye información sobre interacciones proteína-proteína, localizaciones subcelulares, asociaciones con enfermedades, modificaciones postraduccionales, etc.
Programas de visualización molecular (MVS)
PyMOL, Chimera, VMD, Jmol y Blender son MVS que pueden utilizarse para renderizar modelos de estructura computarizada de proteínas. Estos programas ofrecen diversas funciones, como la representación de imágenes y vídeos de alta calidad, el análisis de las interacciones proteína-ligando, etc.
Es posible descargar el archivo de la estructura de la proteína a representar, a través de bases de datos (RCSB PDB, AlphaFold, etc.) u otras herramientas en línea y, a continuación, cargarlo en alguno de los MVS disponibles. Una vez cargado el archivo de estructura, se puede utilizar las herramientas del programa para manipular y visualizar la estructura de la proteína (Imagen 2).
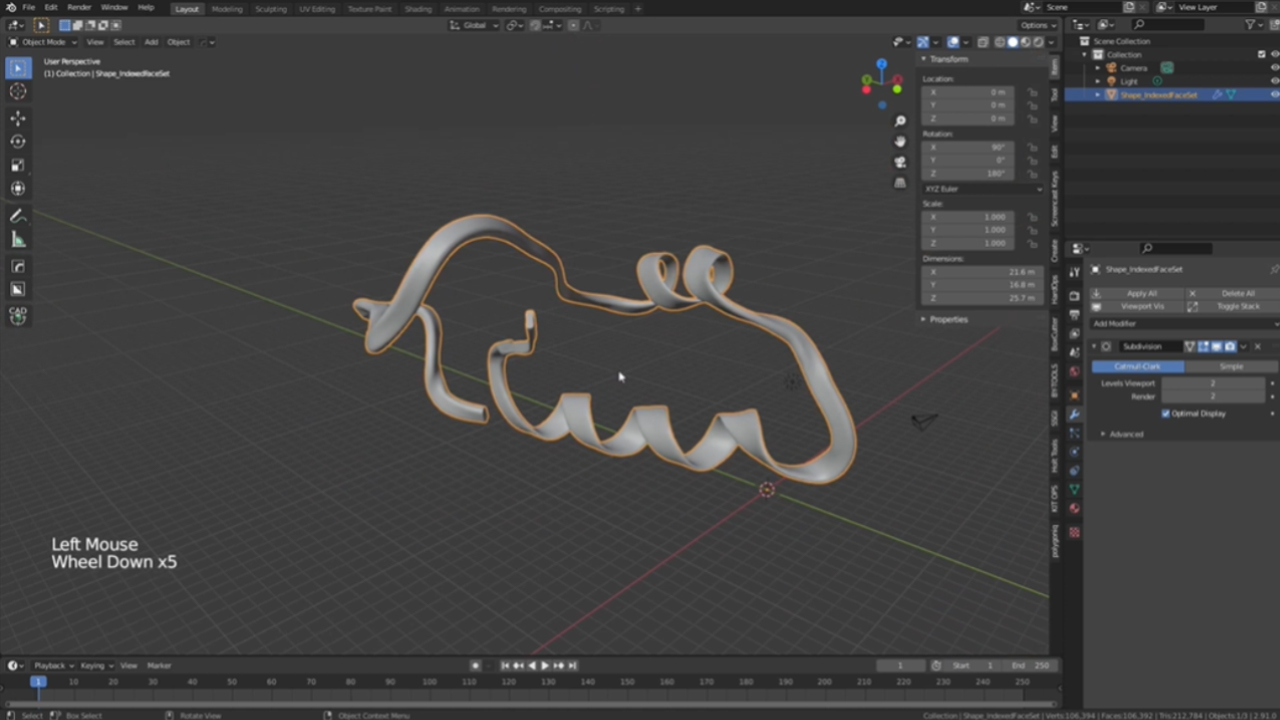
Imagen 2. Visualización 3D de una proteína en Blender.
Una mención especial es para Blender, un software de creación 3D gratuito y de código abierto que soporta la totalidad del pipeline 3D de modelado, aparejado, animación, simulación, renderizado, composición y seguimiento de movimiento, incluso edición de vídeo. Dispone de una gama completa de herramientas de modelado que facilitan la creación, transformación y edición de modelos tridimensionales.
El conjunto de herramientas de modelado de Blender es amplio e incluye compatibilidad total con N-Gon, deslizamiento de bordes, inserción, cuadrícula y relleno de puentes, herramientas y pinceles avanzados para esculpir, subdivisión dinámica y multirresolución, pintura 3D con pinceles texturizados y enmascaramiento. Blender también cuenta con un trazador de rutas de producción de alta gama llamado Cycles que permite crear renderizados de alta calidad.
La elección entre Blender, PyMOL, Chimera, VMD o Jmol depende de las necesidades y preferencias específicas de cada caso. A menudo se prefiere PyMOL para visualizar y crear imágenes con calidad de publicación. VMD es también una opción popular para analizar los resultados de simulaciones de dinámica molecular. Chimera puede utilizarse para el modelado interactivo de homologías.
El plugin de Blender BlendMol es capaz de importar escenas de VMD o PyMOL a Blender y puede utilizarse para crear imágenes mejoradas para su uso en revistas, medios de comunicación, sitios web y clases.
Para finalizar, es importante que pruebe la totalidad de estas herramientas, en búsqueda de la que mejor se adapta a las necesidades de cada proyecto. La mayoría de las veces se empleará más de una herramienta, al no existir un solo programa que contenga todas las características del conjunto.
Artículos relacionados
